Mikrobiologiska provtagningsplaner och kriterier ... - Svensk Mjölk
Mikrobiologiska provtagningsplaner och kriterier ... - Svensk Mjölk
Mikrobiologiska provtagningsplaner och kriterier ... - Svensk Mjölk
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Figur 6 OC-kurvor för olika antal delprov (n) <strong>och</strong> olika <strong>kriterier</strong> för acceptans (c) för tvåklassplaner.<br />
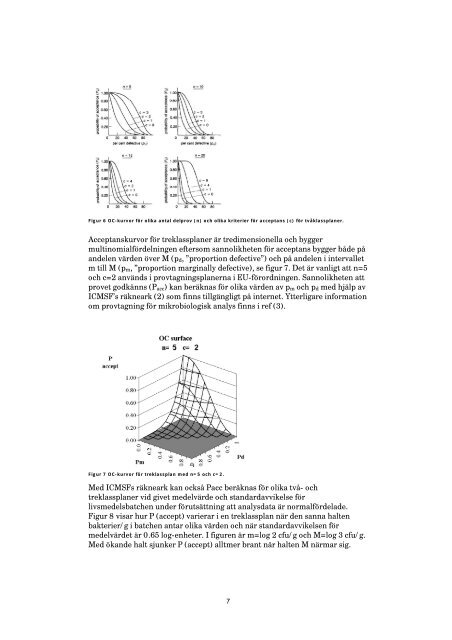
Acceptanskurvor för treklassplaner är tredimensionella <strong>och</strong> bygger<br />
multinomialfördelningen eftersom sannolikheten för acceptans bygger både på<br />
andelen värden över M (pd, ”proportion defective”) <strong>och</strong> på andelen i intervallet<br />
m till M (pm, ”proportion marginally defective), se figur 7. Det är vanligt att n=5<br />
<strong>och</strong> c=2 används i <strong>provtagningsplaner</strong>na i EU-förordningen. Sannolikheten att<br />
provet godkänns (Pacc) kan beräknas för olika värden av pm <strong>och</strong> pd med hjälp av<br />
ICMSF’s räkneark (2) som finns tillgängligt på internet. Ytterligare information<br />
om provtagning för mikrobiologisk analys finns i ref (3).<br />
Figur 7 OC-kurvor för treklassplan med n=5 <strong>och</strong> c=2.<br />
Med ICMSFs räkneark kan också Pacc beräknas för olika två- <strong>och</strong><br />
treklassplaner vid givet medelvärde <strong>och</strong> standardavvikelse för<br />
livsmedelsbatchen under förutsättning att analysdata är normalfördelade.<br />
Figur 8 visar hur P (accept) varierar i en treklassplan när den sanna halten<br />
bakterier/g i batchen antar olika värden <strong>och</strong> när standardavvikelsen för<br />
medelvärdet är 0.65 log-enheter. I figuren är m=log 2 cfu/g <strong>och</strong> M=log 3 cfu/g.<br />
Med ökande halt sjunker P (accept) alltmer brant när halten M närmar sig.<br />
7