Reacción en cadena de la polimerasa (PCR) - FBMC
Reacción en cadena de la polimerasa (PCR) - FBMC Reacción en cadena de la polimerasa (PCR) - FBMC
Genómica Aplicada Verano 2012 gramas con demasiado ruido deben ser eliminados del análisis. Para evaluar la cantidad de ruido, utilizar el valor de m/z: 355. El valor del ruido debe verse en la zona que no presenta picos y generalmente tiene una intensidad alrededor de 100. Recordar este valor. 18.5) Otra cosa importante a tener en cuenta son los picos de los estándares de tiempo de retención (RT): alcanos (m/z: 85) o FAMEs (m/z: 87) (estos últimos son los usados en este TP). Estos compuestos se utilizan para calcular el índice de retención (es decir el tiempo de retención de un compuesto dentro de la columna cromatográfica corregido o estandarizado) de cada pico, ya que este dato es necesario a posteriori para la identificación del compuesto correspondiente. Evaluar que todos los estándares incorporados estén presentes en el cromatograma y la forma de los picos. En este punto también es necesario tomar nota de los tiempos de inicio y fin de cada uno de los estándares, es decir la amplitud de la curva, y la intensidad máxima. Esta información la utilizaremos más adelante para calcular los índices de retención (RI) de cada pico. Es importante considerar la variabilidad que existe a lo largo de la corrida, por ello es conveniente considerar la amplitud de los picos estándares en distintos cromatogramas. 19) Procesamiento de los datos El paso siguiente es procesar la información contenida en los cromatogramas que ahora se encuentra en forma de archivo .cdf. Esta tarea se realiza con el programa TagFinder, brevemente este es un software desarrollado para PC que opera en lenguaje Java creando distintos espacios de trabajo. En primer lugar el programa “busca” los picos dentro de los archivos .cdf y convierte la información de los espectros en archivos .txt. Los datos son importados y “sincronizados”, para ello se identifican los estándares de RT y se calcula el RI de todos los picos. Toda esta información es utilizada por el programa para buscar “mass spectral tags” o simplemente “tags” (marcas espectrales que „parecen‟ compuestos químicos) y generar una matriz de datos con los resultados hallados. Finalmente los tags son comparados con bibliotecas de metabolitos para identificarlos; si bien el resultado de esta comparación lo ofrece el programa, la identificación final de los compuestos se realiza en forma manual mediante la evaluación de los espectros. A continuación detallamos una guía práctica de uso para el análisis de los cromatogramas obtenidos en nuestro TP; la información teórica de los comandos utilizados, así como otras implementaciones del programa pueden consultarse en Luedemann et al (2008). 19.1) Creación de un nuevo espacio de trabajo (workspace). Para comenzar a trabajar en el TagFinder es necesario crear un nuevo espacio de trabajo (wsp), que defina los parámetros de nuestro análisis. Para ello crear una carpeta con el nombre de usuario dentro de la carpeta del TagFinder. 19.2) Abrir el TagFinder, en el menú TAG FINDER, seleccionar la opción “create new workspace” (o bien presio- nar el icono que tiene una estrellita ). Se abre un cuadro que permite definir los parámetros del nuevo espacio de trabajo Crear el wsp en la carpeta correspondiente a cada usuario 20) Búsqueda de picos (obtención de archivos .txt) 20.1) Dentro del wsp abierto, ir al menú TOOLS, seleccionar “external tools” (o bien presionar el ícono que tiene el enchufe ). Se abre un cuadro de diálogo Jar Browser seleccionar “select Jar file”, luego elegir “tagtools” open!. Se despliega una lista de herramientas: Seleccionar “cdftools.PeakFinder” Run!.Pregunta si estamos seguros Yes! Define el número de decimales del workspace, depende de los estándares de RT utilizados: 0 para FAMEs, 2 para Alcanos Define el rango de masas a analizar, los valores 70-600 incluyen todas las masas de interés en este tipo de análisis Elegir el directorio donde se va a crear el wsp, p 28
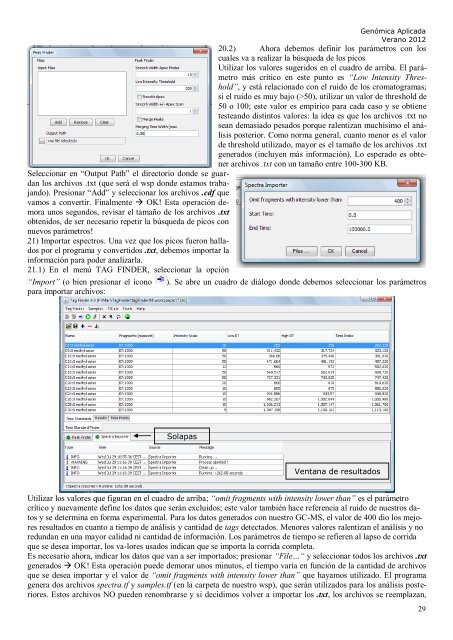
Genómica Aplicada Verano 2012 20.2) Ahora debemos definir los parámetros con los cuales va a realizar la búsqueda de los picos Utilizar los valores sugeridos en el cuadro de arriba. El parámetro más crítico en este punto es “Low Intensity Threshold”, y está relacionado con el ruido de los cromatogramas; si el ruido es muy bajo (>50), utilizar un valor de threshold de 50 o 100; este valor es empírico para cada caso y se obtiene testeando distintos valores: la idea es que los archivos .txt no sean demasiado pesados porque ralentizan muchísimo el análisis posterior. Como norma general, cuanto menor es el valor de threshold utilizado, mayor es el tamaño de los archivos .txt generados (incluyen más información). Lo esperado es obtener archivos .txt con un tamaño entre 100-300 KB. Seleccionar en “Output Path” el directorio donde se guardan los archivos .txt (que será el wsp donde estamos trabajando). Presionar “Add” y seleccionar los archivos .cdf que vamos a convertir. Finalmente OK! Esta operación demora unos segundos, revisar el tamaño de los archivos .txt obtenidos, de ser necesario repetir la búsqueda de picos con nuevos parámetros! 21) Importar espectros. Una vez que los picos fueron hallados por el programa y convertidos .txt, debemos importar la información para poder analizarla. 21.1) En el menú TAG FINDER, seleccionar la opción “Import” (o bien presionar el ícono ). Se abre un cuadro de diálogo donde debemos seleccionar los parámetros para importar archivos: Solapas Ventana de resultados Utilizar los valores que figuran en el cuadro de arriba; “omit fragments with intensity lower than” es el parámetro crítico y nuevamente define los datos que serán excluidos; este valor también hace referencia al ruido de nuestros datos y se determina en forma experimental. Para los datos generados con nuestro GC-MS, el valor de 400 dio los mejores resultados en cuanto a tiempo de análisis y cantidad de tags detectados. Menores valores ralentizan el análisis y no redundan en una mayor calidad ni cantidad de información. Los parámetros de tiempo se refieren al lapso de corrida que se desea importar, los va-lores usados indican que se importa la corrida completa. Es necesario ahora, indicar los datos que van a ser importados; presionar “File…“ y seleccionar todos los archivos .txt generados OK! Esta operación puede demorar unos minutos, el tiempo varía en función de la cantidad de archivos que se desea importar y el valor de “omit fragments with intensity lower than” que hayamos utilizado. El programa genera dos archivos spectra.tf y samples.tf (en la carpeta de nuestro wsp), que serán utilizados para los análisis posteriores. Estos archivos NO pueden renombrarse y si decidimos volver a importar los .txt, los archivos se reemplazan, 29
- Page 1 and 2: Genómica Aplicada Guía de Trabajo
- Page 3 and 4: TP 1: Búsqueda bioinformática de
- Page 5 and 6: de pares de bases depositadas en Ge
- Page 7 and 8: 4) Electroforesis capilar en secuen
- Page 9 and 10: Identificación y caracterización
- Page 11 and 12: e. Comparar los motivos conservados
- Page 13 and 14: ATAATCTCAATAAATATTTGGATGGAAATCGTGTA
- Page 15 and 16: ían ingresado desde Venezuela o di
- Page 17 and 18: vas pérdidas de ADN (RD1- 14). El
- Page 19 and 20: Genómica Aplicada Verano 2012 El t
- Page 21 and 22: Genómica Aplicada Verano 2012 son
- Page 23 and 24: Genómica Aplicada Verano 2012 El m
- Page 25 and 26: Genómica Aplicada Verano 2012 La d
- Page 27: Genómica Aplicada Verano 2012 5) T
- Page 31 and 32: Genómica Aplicada Verano 2012 na 1
- Page 33 and 34: “Results” podremos ver cada met
- Page 35 and 36: Genómica Aplicada Verano 2012 Apre
- Page 37 and 38: 6) Clickear sobre el nombre del pri
- Page 39 and 40: Genómica Aplicada Verano 2012 (may
G<strong>en</strong>ómica Aplicada<br />
Verano 2012<br />
20.2) Ahora <strong>de</strong>bemos <strong>de</strong>finir los parámetros con los<br />
cuales va a realizar <strong>la</strong> búsqueda <strong>de</strong> los picos<br />
Utilizar los valores sugeridos <strong>en</strong> el cuadro <strong>de</strong> arriba. El parámetro<br />
más crítico <strong>en</strong> este punto es “Low Int<strong>en</strong>sity Threshold”,<br />
y está re<strong>la</strong>cionado con el ruido <strong>de</strong> los cromatogramas;<br />
si el ruido es muy bajo (>50), utilizar un valor <strong>de</strong> threshold <strong>de</strong><br />
50 o 100; este valor es empírico para cada caso y se obti<strong>en</strong>e<br />
testeando distintos valores: <strong>la</strong> i<strong>de</strong>a es que los archivos .txt no<br />
sean <strong>de</strong>masiado pesados porque ral<strong>en</strong>tizan muchísimo el análisis<br />
posterior. Como norma g<strong>en</strong>eral, cuanto m<strong>en</strong>or es el valor<br />
<strong>de</strong> threshold utilizado, mayor es el tamaño <strong>de</strong> los archivos .txt<br />
g<strong>en</strong>erados (incluy<strong>en</strong> más información). Lo esperado es obt<strong>en</strong>er<br />
archivos .txt con un tamaño <strong>en</strong>tre 100-300 KB.<br />
Seleccionar <strong>en</strong> “Output Path” el directorio don<strong>de</strong> se guardan<br />
los archivos .txt (que será el wsp don<strong>de</strong> estamos trabajando).<br />
Presionar “Add” y seleccionar los archivos .cdf que<br />
vamos a convertir. Finalm<strong>en</strong>te OK! Esta operación <strong>de</strong>mora<br />
unos segundos, revisar el tamaño <strong>de</strong> los archivos .txt<br />
obt<strong>en</strong>idos, <strong>de</strong> ser necesario repetir <strong>la</strong> búsqueda <strong>de</strong> picos con<br />
nuevos parámetros!<br />
21) Importar espectros. Una vez que los picos fueron hal<strong>la</strong>dos<br />
por el programa y convertidos .txt, <strong>de</strong>bemos importar <strong>la</strong><br />
información para po<strong>de</strong>r analizar<strong>la</strong>.<br />
21.1) En el m<strong>en</strong>ú TAG FINDER, seleccionar <strong>la</strong> opción<br />
“Import” (o bi<strong>en</strong> presionar el ícono ). Se abre un cuadro <strong>de</strong> diálogo don<strong>de</strong> <strong>de</strong>bemos seleccionar los parámetros<br />
para importar archivos:<br />
So<strong>la</strong>pas<br />
V<strong>en</strong>tana <strong>de</strong> resultados<br />
Utilizar los valores que figuran <strong>en</strong> el cuadro <strong>de</strong> arriba; “omit fragm<strong>en</strong>ts with int<strong>en</strong>sity lower than” es el parámetro<br />
crítico y nuevam<strong>en</strong>te <strong>de</strong>fine los datos que serán excluidos; este valor también hace refer<strong>en</strong>cia al ruido <strong>de</strong> nuestros datos<br />
y se <strong>de</strong>termina <strong>en</strong> forma experim<strong>en</strong>tal. Para los datos g<strong>en</strong>erados con nuestro GC-MS, el valor <strong>de</strong> 400 dio los mejores<br />
resultados <strong>en</strong> cuanto a tiempo <strong>de</strong> análisis y cantidad <strong>de</strong> tags <strong>de</strong>tectados. M<strong>en</strong>ores valores ral<strong>en</strong>tizan el análisis y no<br />
redundan <strong>en</strong> una mayor calidad ni cantidad <strong>de</strong> información. Los parámetros <strong>de</strong> tiempo se refier<strong>en</strong> al <strong>la</strong>pso <strong>de</strong> corrida<br />
que se <strong>de</strong>sea importar, los va-lores usados indican que se importa <strong>la</strong> corrida completa.<br />
Es necesario ahora, indicar los datos que van a ser importados; presionar “File…“ y seleccionar todos los archivos .txt<br />
g<strong>en</strong>erados OK! Esta operación pue<strong>de</strong> <strong>de</strong>morar unos minutos, el tiempo varía <strong>en</strong> función <strong>de</strong> <strong>la</strong> cantidad <strong>de</strong> archivos<br />
que se <strong>de</strong>sea importar y el valor <strong>de</strong> “omit fragm<strong>en</strong>ts with int<strong>en</strong>sity lower than” que hayamos utilizado. El programa<br />
g<strong>en</strong>era dos archivos spectra.tf y samples.tf (<strong>en</strong> <strong>la</strong> carpeta <strong>de</strong> nuestro wsp), que serán utilizados para los análisis posteriores.<br />
Estos archivos NO pued<strong>en</strong> r<strong>en</strong>ombrarse y si <strong>de</strong>cidimos volver a importar los .txt, los archivos se reemp<strong>la</strong>zan,<br />
29