Reacción en cadena de la polimerasa (PCR) - FBMC
Reacción en cadena de la polimerasa (PCR) - FBMC
Reacción en cadena de la polimerasa (PCR) - FBMC
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
G<strong>en</strong>ómica Aplicada<br />
Verano 2012<br />
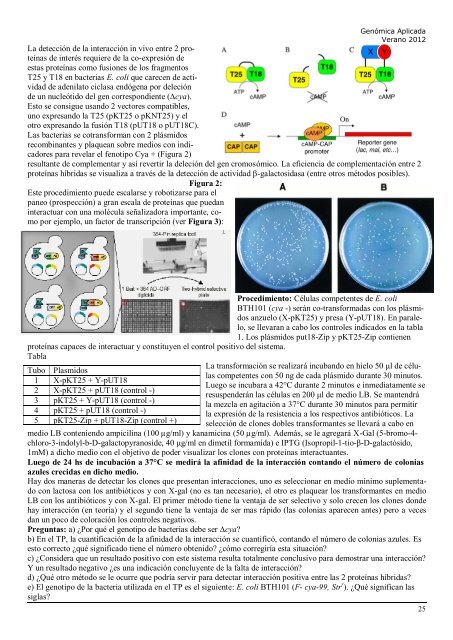
La <strong>de</strong>tección <strong>de</strong> <strong>la</strong> interacción in vivo <strong>en</strong>tre 2 proteínas<br />
<strong>de</strong> interés requiere <strong>de</strong> <strong>la</strong> co-expresión <strong>de</strong><br />
estas proteínas como fusiones <strong>de</strong> los fragm<strong>en</strong>tos<br />
T25 y T18 <strong>en</strong> bacterias E. coli que carec<strong>en</strong> <strong>de</strong> actividad<br />
<strong>de</strong> ad<strong>en</strong>i<strong>la</strong>to cic<strong>la</strong>sa <strong>en</strong>dóg<strong>en</strong>a por <strong>de</strong>leción<br />
<strong>de</strong> un nucleótido <strong>de</strong>l g<strong>en</strong> correspondi<strong>en</strong>te (∆cya).<br />
Esto se consigue usando 2 vectores compatibles,<br />
uno expresando <strong>la</strong> T25 (pKT25 o pKNT25) y el<br />
otro expresando <strong>la</strong> fusión T18 (pUT18 o pUT18C).<br />
Las bacterias se cotransforman con 2 plásmidos<br />
recombinantes y p<strong>la</strong>quean sobre medios con indicadores<br />
para reve<strong>la</strong>r el f<strong>en</strong>otipo Cya + (Figura 2)<br />
resultante <strong>de</strong> complem<strong>en</strong>tar y así revertir <strong>la</strong> <strong>de</strong>leción <strong>de</strong>l g<strong>en</strong> cromosómico. La efici<strong>en</strong>cia <strong>de</strong> complem<strong>en</strong>tación <strong>en</strong>tre 2<br />
proteínas híbridas se visualiza a través <strong>de</strong> <strong>la</strong> <strong>de</strong>tección <strong>de</strong> actividad β-ga<strong>la</strong>ctosidasa (<strong>en</strong>tre otros métodos posibles).<br />
Figura 2:<br />
Este procedimi<strong>en</strong>to pue<strong>de</strong> esca<strong>la</strong>rse y robotizarse para el<br />
paneo (prospección) a gran esca<strong>la</strong> <strong>de</strong> proteínas que puedan<br />
interactuar con una molécu<strong>la</strong> señalizadora importante, como<br />
por ejemplo, un factor <strong>de</strong> transcripción (ver Figura 3):<br />
Procedimi<strong>en</strong>to: Célu<strong>la</strong>s compet<strong>en</strong>tes <strong>de</strong> E. coli<br />
BTH101 (cya -) serán co-transformadas con los plásmidos<br />
anzuelo (X-pKT25) y presa (Y-pUT18). En paralelo,<br />
se llevaran a cabo los controles indicados <strong>en</strong> <strong>la</strong> tab<strong>la</strong><br />
1. Los plásmidos put18-Zip y pKT25-Zip conti<strong>en</strong><strong>en</strong><br />
proteínas capaces <strong>de</strong> interactuar y constituy<strong>en</strong> el control positivo <strong>de</strong>l sistema.<br />
Tab<strong>la</strong><br />
Tubo P<strong>la</strong>smidos<br />
1 X-pKT25 + Y-pUT18<br />
2 X-pKT25 + pUT18 (control -)<br />
3 pKT25 + Y-pUT18 (control -)<br />
4 pKT25 + pUT18 (control -)<br />
5 pKT25-Zip + pUT18-Zip (control +)<br />
La transformación se realizará incubando <strong>en</strong> hielo 50 μl <strong>de</strong> célu<strong>la</strong>s<br />
compet<strong>en</strong>tes con 50 ng <strong>de</strong> cada plásmido durante 30 minutos.<br />
Luego se incubara a 42°C durante 2 minutos e inmediatam<strong>en</strong>te se<br />
resusp<strong>en</strong><strong>de</strong>rán <strong>la</strong>s célu<strong>la</strong>s <strong>en</strong> 200 μl <strong>de</strong> medio LB. Se mant<strong>en</strong>drá<br />
<strong>la</strong> mezc<strong>la</strong> <strong>en</strong> agitación a 37°C durante 30 minutos para permitir<br />
<strong>la</strong> expresión <strong>de</strong> <strong>la</strong> resist<strong>en</strong>cia a los respectivos antibióticos. La<br />
selección <strong>de</strong> clones dobles transformantes se llevará a cabo <strong>en</strong><br />
medio LB cont<strong>en</strong>i<strong>en</strong>do ampicilina (100 µg/ml) y kanamicina (50 µg/ml). A<strong>de</strong>más, se le agregará X-Gal (5-bromo-4chloro-3-indolyl-b-D-ga<strong>la</strong>ctopyranosi<strong>de</strong>,<br />
40 μg/ml <strong>en</strong> dimetil formamida) e IPTG (Isopropil-1-tio-β-D-ga<strong>la</strong>ctósido,<br />
1mM) a dicho medio con el objetivo <strong>de</strong> po<strong>de</strong>r visualizar los clones con proteínas interactuantes.<br />
Luego <strong>de</strong> 24 hs <strong>de</strong> incubación a 37°C se medirá <strong>la</strong> afinidad <strong>de</strong> <strong>la</strong> interacción contando el número <strong>de</strong> colonias<br />
azules crecidas <strong>en</strong> dicho medio.<br />
Hay dos maneras <strong>de</strong> <strong>de</strong>tectar los clones que pres<strong>en</strong>tan interacciones, uno es seleccionar <strong>en</strong> medio mínimo suplem<strong>en</strong>tado<br />
con <strong>la</strong>ctosa con los antibióticos y con X-gal (no es tan necesario), el otro es p<strong>la</strong>quear los transformantes <strong>en</strong> medio<br />
LB con los antibióticos y con X-gal. El primer método ti<strong>en</strong>e <strong>la</strong> v<strong>en</strong>taja <strong>de</strong> ser selectivo y solo crec<strong>en</strong> los clones don<strong>de</strong><br />
hay interacción (<strong>en</strong> teoría) y el segundo ti<strong>en</strong>e <strong>la</strong> v<strong>en</strong>taja <strong>de</strong> ser mas rápido (<strong>la</strong>s colonias aparec<strong>en</strong> antes) pero a veces<br />
dan un poco <strong>de</strong> coloración los controles negativos.<br />
Preguntas: a) ¿Por qué el g<strong>en</strong>otipo <strong>de</strong> bacterias <strong>de</strong>be ser ∆cya?<br />
b) En el TP, <strong>la</strong> cuantificación <strong>de</strong> <strong>la</strong> afinidad <strong>de</strong> <strong>la</strong> interacción se cuantificó, contando el número <strong>de</strong> colonias azules. Es<br />
esto correcto ¿qué significado ti<strong>en</strong>e el número obt<strong>en</strong>ido? ¿cómo corregiría esta situación?<br />
c) ¿Consi<strong>de</strong>ra que un resultado positivo con este sistema resulta totalm<strong>en</strong>te conclusivo para <strong>de</strong>mostrar una interacción?<br />
Y un resultado negativo ¿es una indicación concluy<strong>en</strong>te <strong>de</strong> <strong>la</strong> falta <strong>de</strong> interacción?<br />
d) ¿Qué otro método se le ocurre que podría servir para <strong>de</strong>tectar interacción positiva <strong>en</strong>tre <strong>la</strong>s 2 proteínas híbridas?<br />
e) El g<strong>en</strong>otipo <strong>de</strong> <strong>la</strong> bacteria utilizada <strong>en</strong> el TP es el sigui<strong>en</strong>te: E. coli BTH101 (F- cya-99, Str r ). ¿Qué significan <strong>la</strong>s<br />
sig<strong>la</strong>s?<br />
25