Genetische Untersuchung der Populationsstruktur ... - Die Schmellers
Genetische Untersuchung der Populationsstruktur ... - Die Schmellers
Genetische Untersuchung der Populationsstruktur ... - Die Schmellers
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
72<br />
7.2 POPULATIONSGENETIK<br />
Für die Berechnung des FST-Wertes zwischen Gebieten wurden die Populationen eines<br />
Gebietes ausgewählt, die den geringsten FST-Wert zu allen an<strong>der</strong>en Populationen<br />
dieses Gebietes aufwiesen (siehe Tabellen 7.2-9 und 11.1.10). <strong>Die</strong>se Populationen<br />
zeigen demnach eine von allen an<strong>der</strong>en Populationen eines Gebietes wenig<br />
abweichende genetische Struktur.<br />
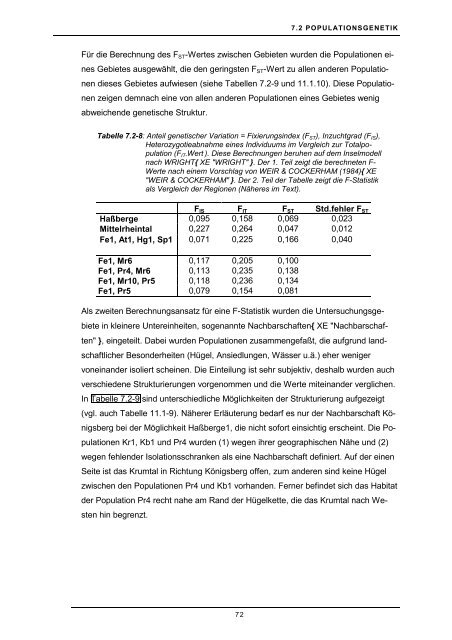
Tabelle 7.2-8: Anteil genetischer Variation = Fixierungsindex (FST), Inzuchtgrad (FIS),<br />
Heterozygotieabnahme eines Individuums im Vergleich zur Totalpopulation<br />
(FIT-Wert ). <strong>Die</strong>se Berechnungen beruhen auf dem Inselmodell<br />
nach WRIGHT{ XE "WRIGHT" }. Der 1. Teil zeigt die berechneten F-<br />
Werte nach einem Vorschlag von WEIR & COCKERHAM (1984){ XE<br />
"WEIR & COCKERHAM" }. Der 2. Teil <strong>der</strong> Tabelle zeigt die F-Statistik<br />
als Vergleich <strong>der</strong> Regionen (Näheres im Text).<br />
FIS FIT FST Std.fehler FST<br />
Haßberge 0,095 0,158 0,069 0,023<br />
Mittelrheintal 0,227 0,264 0,047 0,012<br />
Fe1, At1, Hg1, Sp1 0,071 0,225 0,166 0,040<br />
Fe1, Mr6 0,117 0,205 0,100<br />
Fe1, Pr4, Mr6 0,113 0,235 0,138<br />
Fe1, Mr10, Pr5 0,118 0,236 0,134<br />
Fe1, Pr5 0,079 0,154 0,081<br />
Als zweiten Berechnungsansatz für eine F-Statistik wurden die <strong>Untersuchung</strong>sgebiete<br />
in kleinere Untereinheiten, sogenannte Nachbarschaften{ XE "Nachbarschaften"<br />
}, eingeteilt. Dabei wurden Populationen zusammengefaßt, die aufgrund landschaftlicher<br />
Beson<strong>der</strong>heiten (Hügel, Ansiedlungen, Wässer u.ä.) eher weniger<br />
voneinan<strong>der</strong> isoliert scheinen. <strong>Die</strong> Einteilung ist sehr subjektiv, deshalb wurden auch<br />
verschiedene Strukturierungen vorgenommen und die Werte miteinan<strong>der</strong> verglichen.<br />
In Tabelle 7.2-9 sind unterschiedliche Möglichkeiten <strong>der</strong> Strukturierung aufgezeigt<br />
(vgl. auch Tabelle 11.1-9). Näherer Erläuterung bedarf es nur <strong>der</strong> Nachbarschaft Königsberg<br />
bei <strong>der</strong> Möglichkeit Haßberge1, die nicht sofort einsichtig erscheint. <strong>Die</strong> Populationen<br />
Kr1, Kb1 und Pr4 wurden (1) wegen ihrer geographischen Nähe und (2)<br />
wegen fehlen<strong>der</strong> Isolationsschranken als eine Nachbarschaft definiert. Auf <strong>der</strong> einen<br />
Seite ist das Krumtal in Richtung Königsberg offen, zum an<strong>der</strong>en sind keine Hügel<br />
zwischen den Populationen Pr4 und Kb1 vorhanden. Ferner befindet sich das Habitat<br />
<strong>der</strong> Population Pr4 recht nahe am Rand <strong>der</strong> Hügelkette, die das Krumtal nach Westen<br />
hin begrenzt.