Molekulare Methoden in der mikrobiologischen ... - BIOspektrum
Molekulare Methoden in der mikrobiologischen ... - BIOspektrum
Molekulare Methoden in der mikrobiologischen ... - BIOspektrum
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
744 WISSENSCHAFT · SPECIAL: MOLEKULARE DIAGNOSTIK<br />
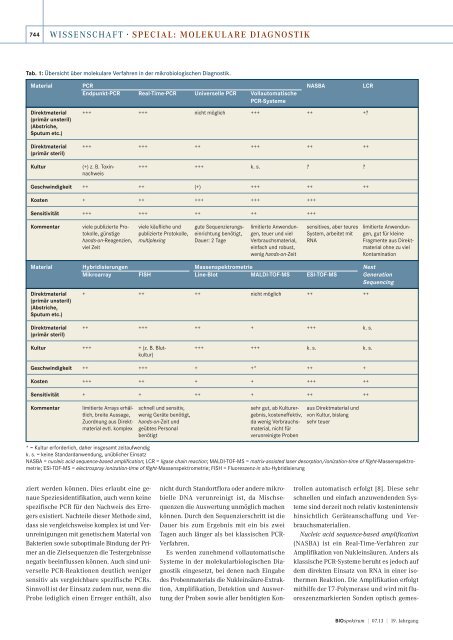
Tab. 1: Übersicht über molekulare Verfahren <strong>in</strong> <strong>der</strong> <strong>mikrobiologischen</strong> Diagnostik.<br />
Material PCR NASBA LCR<br />
Endpunkt-PCR Real-Time-PCR Universelle PCR Vollautomatische<br />
PCR-Systeme<br />
Direktmaterial +++ +++ nicht möglich +++ ++ +?<br />
(primär unsteril)<br />
(Abstriche,<br />
Sputum etc.)<br />
Direktmaterial +++ +++ ++ +++ ++ ++<br />
(primär steril)<br />
Kultur (+) z. B. Tox<strong>in</strong>- +++ +++ k. s. ? ?<br />
nachweis<br />
Geschw<strong>in</strong>digkeit ++ ++ (+) +++ ++ ++<br />
Kosten + ++ +++ +++ +++<br />
Sensitivität +++ +++ ++ ++ +++<br />
Kommentar viele publizierte Pro- viele käufliche und gute Sequenzierungs- limitierte Anwendun- sensitives, aber teures limitierte Anwenduntokolle,<br />
günstige publizierte Protokolle, e<strong>in</strong>richtung benötigt, gen, teuer und viel System, arbeitet mit gen, gut für kle<strong>in</strong>e<br />
hands-on-Reagenzien, multiplex<strong>in</strong>g Dauer: 2 Tage Verbrauchsmaterial, RNA Fragmente aus Direktviel<br />
Zeit e<strong>in</strong>fach und robust, material ohne zu viel<br />
wenig hands-on-Zeit<br />
Kontam<strong>in</strong>ation<br />
Material Hybridisierungen Massenspektrometrie Next<br />
Mikroarray FISH L<strong>in</strong>e-Blot MALDI-TOF-MS ESI-TOF-MS Generation<br />
Sequenc<strong>in</strong>g<br />
Direktmaterial + ++ ++ nicht möglich ++ ++<br />
(primär unsteril)<br />
(Abstriche,<br />
Sputum etc.)<br />
Direktmaterial ++ +++ ++ + +++ k. s.<br />
(primär steril)<br />
Kultur +++ + (z. B. Blut- +++ +++ k. s. k. s.<br />
kultur)<br />
Geschw<strong>in</strong>digkeit ++ +++ + +* ++ +<br />
Kosten +++ ++ + + +++ ++<br />
Sensitivität + + ++ + ++ ++<br />
Kommentar limitierte Arrays erhäl- schnell und sensitiv, sehr gut, ab Kulturer- aus Direktmaterial und<br />
tlich, breite Aussage, wenig Geräte benötigt, gebnis, kosteneffektiv, von Kultur, bislang<br />
Zuordnung aus Direkt- hands-on-Zeit und da wenig Verbrauchs- sehr teuer<br />
material evtl. komplex geübtes Personal material, nicht für<br />
benötigt<br />
verunre<strong>in</strong>igte Proben<br />
* = Kultur erfor<strong>der</strong>lich, daher <strong>in</strong>sgesamt zeitaufwendig<br />
k. s. = ke<strong>in</strong>e Standardanwendung, unüblicher E<strong>in</strong>satz<br />
NASBA = nucleic acid sequence-based amplification; LCR = ligase cha<strong>in</strong> reaction; MALDI-TOF-MS = matrix-assisted laser desorption/ionization-time of flight-Massenspektrometrie;<br />
ESI-TOF-MS = electrospray ionization-time of flight-Massenspektrometrie; FISH = Fluoreszenz-<strong>in</strong> situ-Hybridisierung<br />
ziert werden können. Dies erlaubt e<strong>in</strong>e ge -<br />
naue Spezies identifikation, auch wenn ke<strong>in</strong>e<br />
spezifische PCR für den Nachweis des Erregers<br />
existiert. Nachteile dieser Methode s<strong>in</strong>d,<br />
dass sie vergleichsweise komplex ist und Verunre<strong>in</strong>igungen<br />
mit genetischem Material von<br />
Bakterien sowie suboptimale B<strong>in</strong>dung <strong>der</strong> Primer<br />
an die Zielsequenzen die Testergebnisse<br />
negativ bee<strong>in</strong>flussen können. Auch s<strong>in</strong>d universelle<br />
PCR-Reaktionen deutlich weniger<br />
sensitiv als vergleichbare spezifische PCRs.<br />
S<strong>in</strong>nvoll ist <strong>der</strong> E<strong>in</strong>satz zudem nur, wenn die<br />
Probe lediglich e<strong>in</strong>en Erreger enthält, also<br />
nicht durch Standortflora o<strong>der</strong> an<strong>der</strong>e mikrobielle<br />
DNA verunre<strong>in</strong>igt ist, da Mischsequenzen<br />
die Auswertung unmöglich machen<br />
können. Durch den Sequenzierschritt ist die<br />
Dauer bis zum Ergebnis mit e<strong>in</strong> bis zwei<br />
Tagen auch länger als bei klassischen PCR-<br />
Verfahren.<br />
Es werden zunehmend vollautomatische<br />
Systeme <strong>in</strong> <strong>der</strong> molekularbiologischen Diagnostik<br />
e<strong>in</strong>gesetzt, bei denen nach E<strong>in</strong>gabe<br />
des Probenmaterials die Nukle<strong>in</strong>säure-Extraktion,<br />
Amplifikation, Detektion und Auswertung<br />
<strong>der</strong> Proben sowie aller benötigten Kontrollen<br />
automatisch erfolgt [8]. Diese sehr<br />
schnellen und e<strong>in</strong>fach anzuwendenden Systeme<br />
s<strong>in</strong>d <strong>der</strong>zeit noch relativ kosten<strong>in</strong>tensiv<br />
h<strong>in</strong>sichtlich Geräteanschaffung und Verbrauchsmaterialien.<br />
Nucleic acid sequence-based amplification<br />
(NASBA) ist e<strong>in</strong> Real-Time-Verfahren zur<br />
Amplifikation von Nukle<strong>in</strong>säuren. An<strong>der</strong>s als<br />
klassische PCR-Systeme beruht es jedoch auf<br />
dem direkten E<strong>in</strong>satz von RNA <strong>in</strong> e<strong>in</strong>er isothermen<br />
Reaktion. Die Amplifikation erfolgt<br />
mithilfe <strong>der</strong> T7-Polymerase und wird mit fluoreszenzmarkierten<br />
Sonden optisch gemes-<br />
<strong>BIOspektrum</strong> | 07.13 | 19. Jahrgang