Molekulare Methoden in der mikrobiologischen ... - BIOspektrum
Molekulare Methoden in der mikrobiologischen ... - BIOspektrum
Molekulare Methoden in der mikrobiologischen ... - BIOspektrum
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
743<br />
Nachweis von Infektionserregern<br />
<strong>Molekulare</strong> <strong>Methoden</strong> <strong>in</strong> <strong>der</strong><br />
<strong>mikrobiologischen</strong> Diagnostik<br />
SÖREN SCHUBERT, ANDREAS WIESER<br />
MAX VON PETTENKOFER-INSTITUT FÜR HYGIENE UND MEDIZINISCHE<br />
MIKROBIOLOGIE, LMU MÜNCHEN<br />
Today molecular methods are <strong>in</strong>dispensable <strong>in</strong> diagnostic microbiology.<br />
Several technologies aim to detect pathogens, virulence factor or resistance<br />
genes by identify<strong>in</strong>g specific DNA/RNA sequences. Advantages<br />
compared to culture based methods are the comparatively short time to<br />
results, detection of non-culturable or slow grow<strong>in</strong>g pathogens, and<br />
specific detection of virulence marker genes. Drawbacks <strong>in</strong>clude falsenegative<br />
results due to variation of target structures and the relatively<br />
high costs.<br />
DOI: 10.1007/s12268-013-0386-x<br />
© Spr<strong>in</strong>ger-Verlag 2013<br />
ó Zentrales Ziel <strong>der</strong> <strong>mikrobiologischen</strong> Diagnostik<br />
ist es, <strong>in</strong> Patientenproben Infektionserreger<br />
nachzuweisen und Aussagen zu <strong>der</strong>en<br />
Resistenz gegen antimikrobielle Substanzen<br />
(Antibiotika, Antimykotika) zu machen, um<br />
e<strong>in</strong>e gezielte, effektive Therapie zu ermöglichen.<br />
Von großer Bedeutung ist dabei die<br />
Schnelligkeit. Je früher Informationen über<br />
den Erreger vorliegen, desto eher kann von<br />
e<strong>in</strong>er anfänglich kalkulierten und auf Erfahrungswerten<br />
basierenden Therapie auf e<strong>in</strong>e<br />
gezielte umgestellt werden. Die klassische<br />
mikrobiologische Diagnostik, die auf dem kulturellen<br />
Nachweis von Infektionserregern<br />
beruht, kann dieser For<strong>der</strong>ung nur bed<strong>in</strong>gt<br />
entsprechen. Die Kultur selbst von schnell<br />
wachsenden Bakterien nimmt acht bis zwölf<br />
Stunden <strong>in</strong> Anspruch, an<strong>der</strong>e Infektionserreger<br />
s<strong>in</strong>d schwierig o<strong>der</strong> gar nicht anzüchtbar.<br />
Auch ist <strong>in</strong> e<strong>in</strong>igen Fällen <strong>der</strong> Nachweis<br />
spezifischer Virulenzeigenschaften <strong>der</strong> Erreger<br />
von Interesse. Mit molekularen <strong>Methoden</strong><br />
gel<strong>in</strong>gt es, direkt aus dem Probenmaterial<br />
<strong>in</strong>nerhalb weniger Stunden Infektionserreger<br />
zu identifizieren o<strong>der</strong> spezifische Virulenz-<br />
bzw. Resistenzdeterm<strong>in</strong>anten nachzuweisen<br />
[1–5]. Im Folgenden werden deshalb<br />
die heute zur Verfügung stehenden molekularen<br />
<strong>Methoden</strong> <strong>in</strong> <strong>der</strong> <strong>mikrobiologischen</strong> Diagnostik<br />
kurz vorgestellt (Tab. 1).<br />
<strong>Molekulare</strong> Verfahren <strong>in</strong> <strong>der</strong><br />
<strong>mikrobiologischen</strong> Diagnostik<br />
Der erste Schritt fast aller molekularbiologischen<br />
Diagnostikverfahren ist die Aufre<strong>in</strong>igung<br />
von Nukle<strong>in</strong>säuren aus dem Probenmaterial,<br />
auf welche <strong>der</strong> spezifische Erregernachweis<br />
folgt.<br />
Die Polymerasekettenreaktion (PCR) ist<br />
e<strong>in</strong>e Methode zum spezifischen Nachweis von<br />
Nukle<strong>in</strong>säuren. PCR-basierte Verfahren nutzen<br />
die spezifische komplementäre B<strong>in</strong>dung<br />
kurzer gegenläufiger Oligonukleotide (Primer)<br />
an die nachzuweisende DNA. Bei dieser<br />
Endpunkt-PCR müssen die DNA-Produkte<br />
nach <strong>der</strong> Amplifikation mittels Gelelektrophorese<br />
dargestellt werden. Für die Diagnostik<br />
s<strong>in</strong>d nachfolgende Analysen <strong>der</strong> Produkte<br />
zur Sicherstellung <strong>der</strong> Spezifität erfor<strong>der</strong>lich,<br />
wie Restriktionsverdau o<strong>der</strong> Hybridisierungsverfahren.<br />
Die Endpunkt-PCR ist e<strong>in</strong><br />
relativ schnelles Verfahren, das aufgrund <strong>der</strong><br />
hohen Spezifität und Sensitivität auch aus<br />
Direktmaterial Ergebnisse noch am gleichen<br />
Tag liefern kann. Es besteht außerdem die<br />
Möglichkeit, verschiedene, unterschiedlich<br />
lange Amplifikate parallel im gleichen Ansatz<br />
zu analysieren. Die Gelelektrophorese <strong>der</strong><br />
Produkte und nachfolgende Spezifitätstests<br />
verzögern jedoch die Auswertung und begrenzen<br />
den E<strong>in</strong>satz <strong>in</strong> <strong>der</strong> Rout<strong>in</strong>ediagnostik.<br />
Mo<strong>der</strong>nere PCR-Verfahren nutzen e<strong>in</strong>e<br />
direkte Amplifikationsdetektion, meist mithilfe<br />
e<strong>in</strong>er fluoreszenzmarkierten Sonde. Diese<br />
Real-Time-PCR-Verfahren s<strong>in</strong>d spezifischer<br />
als Endpunkt-PCRs [6]. Ferner s<strong>in</strong>kt <strong>der</strong> Personalzeitaufwand<br />
pro Analyse, da mithilfe <strong>der</strong><br />
Sonde die Amplifikation optisch direkt während<br />
<strong>der</strong> Reaktion gemessen werden kann.<br />
Das macht e<strong>in</strong>e Gelelektrophorese am Ende<br />
<strong>der</strong> Reaktion überflüssig. Die meisten Real-<br />
Time-PCR-Verfahren basieren auf dem E<strong>in</strong>satz<br />
e<strong>in</strong>er mit zwei Farbstoffen, e<strong>in</strong>em<br />
FRET(Förster-Resonanz-Energietransfer)-Paar,<br />
markierten Sonde. E<strong>in</strong> Partner unterdrückt<br />
dabei die Fluoreszenz des an<strong>der</strong>en Farbstoffs,<br />
solange diese räumlich nahe zusammengelagert<br />
s<strong>in</strong>d. Während <strong>der</strong> Amplifikation durch<br />
Abbau <strong>der</strong> Sonde o<strong>der</strong> durch Auflösung e<strong>in</strong>er<br />
Stamm-Loop-Struktur <strong>der</strong> FRET wird diese<br />
räumliche Nähe aufgehoben. Zunehmende<br />
Fluoreszenz<strong>in</strong>tensität repräsentiert daher<br />
e<strong>in</strong>e fortscheitende Amplifikation (Abb. 1).<br />
Alternative Techniken messen nur das Entstehen<br />
größerer Mengen doppelsträngiger<br />
DNA mithilfe <strong>in</strong>terkalieren<strong>der</strong> Farbstoffe. Um<br />
Spezifität sicherzustellen, kann e<strong>in</strong>e Schmelztemperaturkurve<br />
am Ende <strong>der</strong> Reaktion<br />
erstellt werden. Real-Time-PCRs s<strong>in</strong>d sehr<br />
sensitiv und spezifisch und daher sehr gut<br />
geeignet, um Direktmaterial zu untersuchen.<br />
Die amplifizierten DNA-Fragmente und<br />
Zykluszeiten s<strong>in</strong>d recht kurz, die Auswertung<br />
automatisierbar. E<strong>in</strong> Ergebnis ist b<strong>in</strong>nen weniger<br />
Stunden, mit ger<strong>in</strong>gem Arbeitsaufwand<br />
möglich. Diese Vorteile haben dazu geführt,<br />
dass Real-Time-PCR-Verfahren die molekulare<br />
Diagnostik <strong>in</strong> <strong>der</strong> Mikrobiologie dom<strong>in</strong>ieren.<br />
E<strong>in</strong> spezielles PCR-Verfahren <strong>in</strong> <strong>der</strong> <strong>mikrobiologischen</strong><br />
Diagnostik stellen die uni -<br />
versellen PCRs dar, die mit Primern gegen<br />
flankierende, konservierte Bereiche variabler<br />
Sequenzen diese Gene amplifizieren<br />
(16S-rDNA) [7]. Nachfolgend können die variablen<br />
Bereiche sequenziert und mit Datenbanken<br />
abgeglichen werden. Es ist möglich,<br />
die Primer so zu wählen, dass praktisch alle<br />
Bakterien o<strong>der</strong> Pilze detektiert und identifi-<br />
<strong>BIOspektrum</strong> | 07.13 | 19. Jahrgang
744 WISSENSCHAFT · SPECIAL: MOLEKULARE DIAGNOSTIK<br />
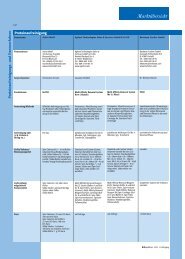
Tab. 1: Übersicht über molekulare Verfahren <strong>in</strong> <strong>der</strong> <strong>mikrobiologischen</strong> Diagnostik.<br />
Material PCR NASBA LCR<br />
Endpunkt-PCR Real-Time-PCR Universelle PCR Vollautomatische<br />
PCR-Systeme<br />
Direktmaterial +++ +++ nicht möglich +++ ++ +?<br />
(primär unsteril)<br />
(Abstriche,<br />
Sputum etc.)<br />
Direktmaterial +++ +++ ++ +++ ++ ++<br />
(primär steril)<br />
Kultur (+) z. B. Tox<strong>in</strong>- +++ +++ k. s. ? ?<br />
nachweis<br />
Geschw<strong>in</strong>digkeit ++ ++ (+) +++ ++ ++<br />
Kosten + ++ +++ +++ +++<br />
Sensitivität +++ +++ ++ ++ +++<br />
Kommentar viele publizierte Pro- viele käufliche und gute Sequenzierungs- limitierte Anwendun- sensitives, aber teures limitierte Anwenduntokolle,<br />
günstige publizierte Protokolle, e<strong>in</strong>richtung benötigt, gen, teuer und viel System, arbeitet mit gen, gut für kle<strong>in</strong>e<br />
hands-on-Reagenzien, multiplex<strong>in</strong>g Dauer: 2 Tage Verbrauchsmaterial, RNA Fragmente aus Direktviel<br />
Zeit e<strong>in</strong>fach und robust, material ohne zu viel<br />
wenig hands-on-Zeit<br />
Kontam<strong>in</strong>ation<br />
Material Hybridisierungen Massenspektrometrie Next<br />
Mikroarray FISH L<strong>in</strong>e-Blot MALDI-TOF-MS ESI-TOF-MS Generation<br />
Sequenc<strong>in</strong>g<br />
Direktmaterial + ++ ++ nicht möglich ++ ++<br />
(primär unsteril)<br />
(Abstriche,<br />
Sputum etc.)<br />
Direktmaterial ++ +++ ++ + +++ k. s.<br />
(primär steril)<br />
Kultur +++ + (z. B. Blut- +++ +++ k. s. k. s.<br />
kultur)<br />
Geschw<strong>in</strong>digkeit ++ +++ + +* ++ +<br />
Kosten +++ ++ + + +++ ++<br />
Sensitivität + + ++ + ++ ++<br />
Kommentar limitierte Arrays erhäl- schnell und sensitiv, sehr gut, ab Kulturer- aus Direktmaterial und<br />
tlich, breite Aussage, wenig Geräte benötigt, gebnis, kosteneffektiv, von Kultur, bislang<br />
Zuordnung aus Direkt- hands-on-Zeit und da wenig Verbrauchs- sehr teuer<br />
material evtl. komplex geübtes Personal material, nicht für<br />
benötigt<br />
verunre<strong>in</strong>igte Proben<br />
* = Kultur erfor<strong>der</strong>lich, daher <strong>in</strong>sgesamt zeitaufwendig<br />
k. s. = ke<strong>in</strong>e Standardanwendung, unüblicher E<strong>in</strong>satz<br />
NASBA = nucleic acid sequence-based amplification; LCR = ligase cha<strong>in</strong> reaction; MALDI-TOF-MS = matrix-assisted laser desorption/ionization-time of flight-Massenspektrometrie;<br />
ESI-TOF-MS = electrospray ionization-time of flight-Massenspektrometrie; FISH = Fluoreszenz-<strong>in</strong> situ-Hybridisierung<br />
ziert werden können. Dies erlaubt e<strong>in</strong>e ge -<br />
naue Spezies identifikation, auch wenn ke<strong>in</strong>e<br />
spezifische PCR für den Nachweis des Erregers<br />
existiert. Nachteile dieser Methode s<strong>in</strong>d,<br />
dass sie vergleichsweise komplex ist und Verunre<strong>in</strong>igungen<br />
mit genetischem Material von<br />
Bakterien sowie suboptimale B<strong>in</strong>dung <strong>der</strong> Primer<br />
an die Zielsequenzen die Testergebnisse<br />
negativ bee<strong>in</strong>flussen können. Auch s<strong>in</strong>d universelle<br />
PCR-Reaktionen deutlich weniger<br />
sensitiv als vergleichbare spezifische PCRs.<br />
S<strong>in</strong>nvoll ist <strong>der</strong> E<strong>in</strong>satz zudem nur, wenn die<br />
Probe lediglich e<strong>in</strong>en Erreger enthält, also<br />
nicht durch Standortflora o<strong>der</strong> an<strong>der</strong>e mikrobielle<br />
DNA verunre<strong>in</strong>igt ist, da Mischsequenzen<br />
die Auswertung unmöglich machen<br />
können. Durch den Sequenzierschritt ist die<br />
Dauer bis zum Ergebnis mit e<strong>in</strong> bis zwei<br />
Tagen auch länger als bei klassischen PCR-<br />
Verfahren.<br />
Es werden zunehmend vollautomatische<br />
Systeme <strong>in</strong> <strong>der</strong> molekularbiologischen Diagnostik<br />
e<strong>in</strong>gesetzt, bei denen nach E<strong>in</strong>gabe<br />
des Probenmaterials die Nukle<strong>in</strong>säure-Extraktion,<br />
Amplifikation, Detektion und Auswertung<br />
<strong>der</strong> Proben sowie aller benötigten Kontrollen<br />
automatisch erfolgt [8]. Diese sehr<br />
schnellen und e<strong>in</strong>fach anzuwendenden Systeme<br />
s<strong>in</strong>d <strong>der</strong>zeit noch relativ kosten<strong>in</strong>tensiv<br />
h<strong>in</strong>sichtlich Geräteanschaffung und Verbrauchsmaterialien.<br />
Nucleic acid sequence-based amplification<br />
(NASBA) ist e<strong>in</strong> Real-Time-Verfahren zur<br />
Amplifikation von Nukle<strong>in</strong>säuren. An<strong>der</strong>s als<br />
klassische PCR-Systeme beruht es jedoch auf<br />
dem direkten E<strong>in</strong>satz von RNA <strong>in</strong> e<strong>in</strong>er isothermen<br />
Reaktion. Die Amplifikation erfolgt<br />
mithilfe <strong>der</strong> T7-Polymerase und wird mit fluoreszenzmarkierten<br />
Sonden optisch gemes-<br />
<strong>BIOspektrum</strong> | 07.13 | 19. Jahrgang
746 WISSENSCHAFT · SPECIAL: MOLEKULARE DIAGNOSTIK<br />
˚ Abb. 1: Real-Time-PCR e<strong>in</strong>er logarithmischen Verdünnungsreihe e<strong>in</strong>er Probe. Die e<strong>in</strong>zelnen Kurven<br />
stellen die jeweilige Fluoreszenzzunahme während <strong>der</strong> Real-Time-PCR und damit die spezifische<br />
DNA-Amplifikation dar. Der CT-Wert ist e<strong>in</strong> Maß für die Menge <strong>der</strong> DNA <strong>in</strong> <strong>der</strong> Ausgangsprobe<br />
und lässt die Bestimmung <strong>der</strong> Erregermenge zu. Niedrige CT-Werte repräsentieren höhere<br />
DNA-Konzentrationen bzw. Erregermengen.<br />
sen. Durch die zyklenlose Amplifikation<br />
und direkte Detektion s<strong>in</strong>d die Analysezeiten<br />
sehr kurz. Allerd<strong>in</strong>gs ist das Verfahren relativ<br />
komplex und durch den primären E<strong>in</strong> -<br />
satz von RNA hauptsächlich zum Nachweis<br />
von RNA-Viren o<strong>der</strong> mRNA-Transkripten<br />
geeignet. Die Instabilität von RNA <strong>in</strong> <strong>der</strong><br />
Probe und die Komplexität <strong>der</strong> relativ teuren<br />
Methode haben e<strong>in</strong>e weitere Verbreitung<br />
beh<strong>in</strong><strong>der</strong>t.<br />
Die Ligasekettenreaktion (ligase cha<strong>in</strong> reaction,<br />
LCR) basiert auf vier komplementären<br />
Nukleotiden als Primer. Die Sequenzen bef<strong>in</strong>den<br />
sich genau nebene<strong>in</strong>an<strong>der</strong> auf <strong>der</strong> Ziel-<br />
DNA, sodass im Falle <strong>der</strong> B<strong>in</strong>dung jeweils<br />
zwei <strong>der</strong> Primer mite<strong>in</strong>an<strong>der</strong> kovalent verbunden<br />
werden. Die an<strong>der</strong>en zwei Primer<br />
s<strong>in</strong>d dazu komplementär und werden im<br />
nächsten Zyklus verbunden, sobald sie nebene<strong>in</strong>an<strong>der</strong><br />
an die <strong>in</strong> vorheriger Reaktion verbundenen<br />
Fragmente b<strong>in</strong>den. Da ke<strong>in</strong>e Amplifikation<br />
erfolgt, s<strong>in</strong>d die Zykluszeiten sehr<br />
kurz. E<strong>in</strong> Problem stellt die Detektion des kurzen<br />
Amplifikats dar. Es werden daher markierte<br />
Oligonukleotide verwendet, <strong>der</strong>en Ligationsprodukte<br />
<strong>in</strong> ELISA-artigen Systemen<br />
detektiert werden können.<br />
Hybridisierungen basieren wie PCR-<br />
Verfahren auf spezifischer komplementärer<br />
B<strong>in</strong>dung zwischen Nukleotidsonden und<br />
Nukle<strong>in</strong>säure-Zielstrukturen. Die Detektion<br />
erfolgt aber ohne enzymatische Nukle<strong>in</strong>säure-Amplifikation.<br />
Die Mikroarray-Technik basiert auf immobilisierten<br />
spezifischen Oligonukleotiden, die<br />
auf e<strong>in</strong>er Festkörperoberfläche fixiert s<strong>in</strong>d.<br />
Die DNA des Probenmaterials wird zunächst<br />
extrahiert, dann amplifiziert und dabei markiert.<br />
Die B<strong>in</strong>dung an die immobilisierten<br />
Sequenzen auf dem Array erfolgt unter möglichst<br />
str<strong>in</strong>genten Bed<strong>in</strong>gungen und ermöglicht<br />
so maximale Spezifität. Nach <strong>der</strong> Hybridisierung<br />
werden automatisiert die Positionen<br />
auf dem Array detektiert, an die die markierten<br />
Amplifikate gebunden haben. Aus <strong>der</strong><br />
Komb<strong>in</strong>ation <strong>der</strong> detektierten DNA-Fragmente<br />
können Rückschlüsse auf die Art des Erregers<br />
o<strong>der</strong> dessen Eigenschaften gezogen werden.<br />
Der Hauptvorteil <strong>der</strong> Mikroarray-Technik<br />
ist, dass e<strong>in</strong>e große Menge spezifischer<br />
Nachweisreaktionen mit überschaubarem<br />
Aufwand parallel durchgeführt werden kann.<br />
Die Kosten für Material und Geräte s<strong>in</strong>d<br />
jedoch relativ hoch.<br />
Fluoreszenz-<strong>in</strong> situ-Hybridisierung (FISH)<br />
ist e<strong>in</strong>e Technik, die ihre Spezifität ebenfalls<br />
aus <strong>der</strong> komplementären Basenpaarung <strong>der</strong><br />
Sonde mit dem Erreger erreicht. Es kann<br />
dabei sowohl DNA als auch RNA nachgewiesen<br />
werden [9]. Voraussetzung für die Hybridisierung<br />
ist die Fixierung und Permeabilisierung<br />
<strong>der</strong> Erreger. Die Untersuchungsprobe<br />
wird mit fluoreszenzmarkierten Sonden <strong>in</strong>kubiert<br />
und fluoreszenzmikroskopisch ausgewertet.<br />
Gemischte Proben und Direktmaterial<br />
können analysiert werden, da sich die e<strong>in</strong>zelnen<br />
Erreger räumlich trennen lassen und<br />
jeweils als positiv o<strong>der</strong> negativ dargestellt<br />
werden können. Um e<strong>in</strong>e ausreichende Signalstärke<br />
für mikroskopische Analysen zu<br />
erreichen, wird meist e<strong>in</strong>e Zielstruktur <strong>in</strong> <strong>der</strong><br />
Zelle gewählt, die mehrfach vorhanden ist,<br />
wie ribosomale RNA (rRNA). Da die Erreger<br />
fixiert werden, ist e<strong>in</strong>e Lebend/Tot-Unterscheidung<br />
kaum möglich. Ausgebildetes Personal<br />
kann Proben b<strong>in</strong>nen e<strong>in</strong>er halben Stunde<br />
ohne großen Materialaufwand vorbereiten<br />
und analysieren.<br />
Die Technik <strong>der</strong> reversen L<strong>in</strong>e-Blot-Hybridisierung<br />
ist dem Mikroarray ähnlich. DNA<br />
o<strong>der</strong> RNA wird aus <strong>der</strong> Probe isoliert, mit<br />
e<strong>in</strong>er PCR-Reaktion amplifiziert und dabei<br />
markiert. Danach wird die entstandene<br />
Mischung aus DNA-Molekülen chemisch <strong>in</strong><br />
e<strong>in</strong>zelsträngige DNA denaturiert und mit<br />
immobilisierter DNA auf <strong>der</strong> Membran des<br />
Blotstreifens hybridisiert. Spezifische B<strong>in</strong>dungen<br />
werden enzymatisch detektiert und<br />
bewirken e<strong>in</strong>en Farbumschlag. So kann e<strong>in</strong>fach<br />
e<strong>in</strong>e Vielzahl an Gensequenzen <strong>in</strong> e<strong>in</strong>er<br />
Probe spezifisch nachgewiesen werden. Es ist<br />
neben <strong>der</strong> Speziesidentifikation auch <strong>der</strong><br />
Nachweis von Resistenzgenen möglich. Der<br />
Arbeitsaufwand ist bei hohen Probenzahlen<br />
vergleichsweise hoch, so eignet sich die<br />
Methode vor allem dort, wo bei wenigen Proben<br />
verschiedene DNA-Zielstrukturen detektiert<br />
werden sollen (z. B. Tuberkulose-Diagnostik).<br />
Die MALDI-TOF(matrix-assisted laser de -<br />
sorption/ionization-time of flight)-Massenspektrometrie<br />
hat sich <strong>in</strong> den letzten Jahren<br />
zur bakteriellen Identifikation durchgesetzt<br />
und an<strong>der</strong>e Verfahren weitgehend verdrängt.<br />
Sie basiert auf <strong>der</strong> Analyse ribosomaler Prote<strong>in</strong>spektren<br />
von Re<strong>in</strong>kulturen und ist von<br />
<strong>der</strong> DNA <strong>der</strong> Erreger unabhängig [10].<br />
Neue MS-basierte diagnostische Systeme<br />
bauen auf e<strong>in</strong>e Komb<strong>in</strong>ation von PCR und<br />
massenspektrometrischer Analyse <strong>der</strong> DNA-<br />
Amplifikate mittels Elektronenspray-Ionisations-time<br />
of flight-Massenspektrometrie (ESI-<br />
TOF-MS). Damit ist e<strong>in</strong>e Bestimmung <strong>der</strong><br />
Sequenz <strong>der</strong> Amplifikate <strong>in</strong> Grenzen möglich.<br />
Die Analysen werden im Mikrotiterplattenformat<br />
durchgeführt und be<strong>in</strong>halten verschiedene<br />
PCR-Reaktionen. E<strong>in</strong> computergestütztes<br />
Auswertesystem kann aus den Komb<strong>in</strong>ationen<br />
<strong>der</strong> Messungen neben <strong>der</strong> Keim -<br />
identifikation auch Stammtypisierungen und<br />
<strong>BIOspektrum</strong> | 07.13 | 19. Jahrgang
747<br />
Tox<strong>in</strong>- sowie Resistenzgen-Nachweise durchführen.<br />
Innerhalb von acht Stunden ist e<strong>in</strong>e<br />
relativ breite Analyse möglich. Das bisherige<br />
System ist extrem teuer und daher für die<br />
meisten diagnostischen Labore ungeeignet.<br />
Next Generation Sequenc<strong>in</strong>g bezeichnet<br />
hochparallele DNA-Sequenzierungen. Es s<strong>in</strong>d<br />
verschiedene Systeme kommerziell verfügbar,<br />
die auf unterschiedlichen Techniken beruhen.<br />
Allen Systemen ist geme<strong>in</strong>, dass Genstücke<br />
aus Direktmaterialien mit o<strong>der</strong> ohne<br />
Amplifikationsschritt sequenziert und mittels<br />
Computeranalyse und Datenbankvergleich<br />
bestimmten Organismen zugeordnet werden.<br />
Es ist nicht nur die Detektion von Spezies,<br />
son<strong>der</strong>n auch von Resistenz- o<strong>der</strong> Virulenzgenen<br />
möglich. Nicht immer ist aus e<strong>in</strong>em<br />
Keimgemisch klar, welche Gensequenzen<br />
(etwa von Plasmiden) zu welchem Erreger<br />
gehören. Auch e<strong>in</strong>e Unterscheidung zwischen<br />
lebenden und toten Erregern o<strong>der</strong> re<strong>in</strong>er DNA<br />
ist nicht möglich. Die Auswertung <strong>der</strong> Daten<br />
ist rechen<strong>in</strong>tensiv. Für manche Fragestellungen,<br />
vor allem bei nicht anzüchtbaren o<strong>der</strong><br />
unbekannten Erregern kann diese aufwendige<br />
Methode <strong>in</strong>teressant se<strong>in</strong>. Neue schnellere<br />
und günstigere Sequenzierungstechniken<br />
machen die Massivsequenzierung zu e<strong>in</strong>em<br />
<strong>in</strong>teressanten Ansatz, <strong>der</strong> <strong>in</strong> den kommenden<br />
Jahren verstärkt experimentell e<strong>in</strong>gesetzt<br />
werden wird und <strong>in</strong> Zukunft e<strong>in</strong>e <strong>in</strong>teressante<br />
diagnostische Alternative bieten könnte [11].<br />
Zusammenfassung und Ausblick<br />
Die molekulare Diagnostik beruht <strong>der</strong>zeit<br />
hauptsächlich auf <strong>der</strong> Analyse von Erbsubstanz<br />
o<strong>der</strong> Prote<strong>in</strong>en <strong>der</strong> Erreger. Der Hauptunterschied<br />
zu klassischen kulturbasierten<br />
Verfahren ist meistens die höhere Geschw<strong>in</strong>digkeit.<br />
Manche Erreger s<strong>in</strong>d gar nicht<br />
anzüchtbar, sodass ihr direkter Nachweis nur<br />
über molekulare Verfahren gel<strong>in</strong>gt. Trotz <strong>der</strong><br />
Vielfalt mo<strong>der</strong>ner molekularer Verfahren ist<br />
die kulturelle Anzucht noch notwendig. Sie<br />
hat meist nicht nur e<strong>in</strong>e höhere Sensitivität,<br />
son<strong>der</strong>n erlaubt auch die e<strong>in</strong>fache Trennung<br />
von Mischkulturen sowie e<strong>in</strong>e Unterscheidung<br />
zwischen toten und lebenden (anzüchtbaren)<br />
Organismen und dies zu meist deutlich<br />
ger<strong>in</strong>geren Kosten. Für e<strong>in</strong>e zuverlässige<br />
Resistenztestung ist außerdem <strong>der</strong> Genotyp<br />
aufgrund <strong>der</strong> Vielfalt an möglichen Resistenzmechanismen<br />
<strong>der</strong> phänotypischen Resistenztestung<br />
unterlegen. Die stete Weiterentwicklung<br />
molekularer Testsysteme zielt darauf,<br />
schnell hochspezifische Resultate zu liefern<br />
und die Tests möglichst e<strong>in</strong>fach durchführbar<br />
zu machen (Abb. 2). Wenn die Kosten<br />
erster Tag zweiter<br />
Tag<br />
dritter Tag<br />
Kulturwachstum<br />
Zeit zur Diagnosestellung<br />
DNA/RNA-<br />
Extraktion<br />
Zwischenschritte<br />
˚ Abb. 2: <strong>Molekulare</strong> Verfahren <strong>der</strong> <strong>mikrobiologischen</strong> Diagnostik und <strong>der</strong>en Zeitaufwand.<br />
dabei für das Labor reduziert werden können,<br />
so werden sich <strong>in</strong> Zukunft molekulare Verfahren<br />
mehr und mehr gegenüber <strong>der</strong> klassischen,<br />
kulturbasierten <strong>mikrobiologischen</strong><br />
Diagnostik durchsetzen.<br />
ó<br />
Literatur<br />
[1] Tenover FC (2011) Develop<strong>in</strong>g molecular amplification<br />
methods for rapid diagnosis of respiratory tract <strong>in</strong>fections<br />
caused by bacterial pathogens.<br />
Cl<strong>in</strong> Infect Dis 52(Suppl 4):S338–345<br />
[2] Cathomas G (2009) Molecular diagnostic <strong>in</strong> <strong>in</strong>fectious<br />
disease pathology: an update. Ann Pathol 29:S19–21<br />
[3] Krishna NK, Cunnion KM (2012) Role of molecular diagnostics<br />
<strong>in</strong> the management of <strong>in</strong>fectious disease emergencies.<br />
Med Cl<strong>in</strong> North Am 96:1067–1078<br />
[4] Balas<strong>in</strong>gham SV, Davidsen T, Szp<strong>in</strong>da I et al. (2009)<br />
Molecular diagnostics <strong>in</strong> tuberculosis: basis and implications<br />
for therapy. Mol Diagn Ther 13:137–151<br />
[5] Bille J (2010) New nonculture-based methods for the diagnosis<br />
of <strong>in</strong>vasive candidiasis.<br />
Curr Op<strong>in</strong> Crit Care 16:460–464<br />
[6] Maur<strong>in</strong> M (2012) Real-time PCR as a diagnostic tool for<br />
bacterial diseases. Expert Rev Mol Diagn 12:731–754<br />
[7] Preuner S, Lion T (2009) Towards molecular diagnostics<br />
of <strong>in</strong>vasive fungal <strong>in</strong>fections.<br />
Expert Rev Mol Diagn 9:397–401<br />
[8] Nolte FS (2008) Molecular diagnostics for detection of<br />
bacterial and viral pathogens <strong>in</strong> community-acquired pneumonia.<br />
Cl<strong>in</strong> Infect Dis 47(Suppl 3):S123–126<br />
AUTOREN<br />
Probennahme<br />
Kulturwachstum<br />
Sören Schubert<br />
Jahrgang 1966. 1985–1992<br />
Mediz<strong>in</strong>studium an <strong>der</strong> Universität<br />
Hamburg. 1993–1995 Dissertation<br />
an <strong>der</strong> Universität<br />
Würzburg. Seit 1998 Arbeitsgruppenleiter<br />
am Max von<br />
Pettenkofer-Institut für Hygiene<br />
und Mediz<strong>in</strong>ische Mikrobiologie<br />
<strong>der</strong> LMU München. 2006 Habilitation.<br />
Seit 2006 leiten<strong>der</strong><br />
Oberarzt <strong>der</strong> Diagnostik am Max<br />
von Pettenkofer-Institut.<br />
FISH<br />
direkt MALDI-TOF (Ur<strong>in</strong>)<br />
automatisierte PCR-Verfahren<br />
PCR-Verfahren, LCR, NASBA<br />
RT-PCR-Verfahren<br />
Massivsequenzierung<br />
ESI-TOF<br />
universelle PCR/Sequenzierung<br />
L<strong>in</strong>e Probe Assay<br />
Mikroarray<br />
MALDI-TOF<br />
FISH von Kultur<br />
PCR von Kultur (RT o<strong>der</strong> konventionell)<br />
an<strong>der</strong>e molekulare Verfahren aus Kultur<br />
[9] Montone KT, Guarner J (2013) In situ hybridization for<br />
rRNA sequences <strong>in</strong> anatomic pathology specimens, applications<br />
for fungal pathogen detection: a review.<br />
Adv Anat Pathol 20:168–174<br />
[10] Wieser A, Schnei<strong>der</strong> L, Jung J et al. (2012) MALDI-TOF<br />
MS <strong>in</strong> microbiological diagnostics-identification of microorganisms<br />
and beyond. Appl Microbiol Biotechnol 93:965–974<br />
[11] Dunne WM Jr, Westblade LF, Ford B (2012) Next-generation<br />
and whole-genome sequenc<strong>in</strong>g <strong>in</strong> the diagnostic cl<strong>in</strong>ical<br />
microbiology laboratory.<br />
Eur J Cl<strong>in</strong> Microbiol Infect Dis 31:1719–1726<br />
Korrespondenzadresse:<br />
Prof. Dr. med. Sören Schubert<br />
Max von Pettenkofer-Institut<br />
Ludwig-Maximilians-Universität München<br />
Marchion<strong>in</strong>istraße 17<br />
D-81377 München<br />
Tel.: 089-2180-78202<br />
Fax: 089-2180-78294<br />
schubert@med.uni-muenchen.de<br />
Andreas Wieser<br />
Jahrgang 1983. 2002–2009<br />
Mediz<strong>in</strong>studium an <strong>der</strong> LMU<br />
München. 2005–2009 Dissertation<br />
am Max von Pettenkofer-Institut<br />
für Hygiene und<br />
Mediz<strong>in</strong>ische Mikrobiologie<br />
<strong>der</strong> LMU München, dort seit<br />
2009 Postdoc <strong>in</strong> <strong>der</strong> Arbeitsgruppe<br />
von Prof. Dr. Schubert.<br />
<strong>BIOspektrum</strong> | 07.13 | 19. Jahrgang