de lëtzebuerger Ziichter 3/2013 - Convis Herdbuch Service Elevage ...
de lëtzebuerger Ziichter 3/2013 - Convis Herdbuch Service Elevage ...
de lëtzebuerger Ziichter 3/2013 - Convis Herdbuch Service Elevage ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
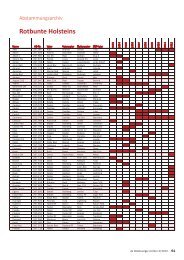
MILCHRINDER 35<br />
EuroGenomics-10K-LD-Chip<br />
Was versteht man unter Imputing?<br />
Dr. Stefan Rensing, vit<br />
Wörtlich übersetzt bezeichnet <strong>de</strong>r<br />
Begriff „imputing“ so viel wie<br />
„Kalkulation/Hochrechnung“.<br />
Im Zusammenhang mit <strong>de</strong>r genomischen<br />
Zuchtwertschätzung be<strong>de</strong>utet er Folgen<strong>de</strong>s:<br />
Aktuell basiert die genomische<br />
Zuchtwertschätzung auf <strong>de</strong>r Typisierung<br />
von 54.000 SNPs (54K-Chip). Wenn diese<br />
SNPs nicht vollständig vorliegen, können<br />
sie mittels eines statistisch-genetischen<br />
Verfahrens berechnet o<strong>de</strong>r besser „vorhergesagt“<br />
wer<strong>de</strong>n. Fehlt nur ein kleiner<br />
Teil <strong>de</strong>r SNPs, weil z.B. im Labor nur 98%<br />
<strong>de</strong>r 54.000 SNPs i<strong>de</strong>ntifiziert wer<strong>de</strong>n<br />
konnten, funktioniert das Imputing mit<br />
nahezu 100% Sicherheit. Die Hauptanwendung<br />
<strong>de</strong>s Verfahrens ist aber die<br />
Hochrechnung von Typisierungen mit <strong>de</strong>m<br />
kostengünstigeren Low-Density-Chip (ab<br />
6K) auf die vollen 54K-SNP. Dabei müssen<br />
bei LD-Typisierungen mit z.B. 6.000<br />
SNP fast 90% <strong>de</strong>r SNP „imputet“ wer<strong>de</strong>n.<br />
Trotz<strong>de</strong>m beträgt die Fehlerrate dabei weniger<br />
als 2%, und auch die anschließend<br />
berechneten genomischen Zuchtwerte<br />
haben je nach Merkmal eine um nur 1<br />
bis 3% niedrigere Sicherheit. Da die Entwicklung<br />
im Bereich <strong>de</strong>r Chips rasant vorangeht<br />
und damit die Vielfalt <strong>de</strong>r jeweils<br />
typisierten SNPs zunimmt, ist das Imputing<br />
künftig fester Bestandteil <strong>de</strong>r genomischen<br />
Zuchtwertschätzung. Nur so können<br />
die Typisierungsergebnisse auf die in<br />
<strong>de</strong>r jeweiligen Lernstichprobe zugrun<strong>de</strong><br />
gelegte Zahl an SNPs (54k = Volltypisierung)<br />
vereinheitlicht wer<strong>de</strong>n.<br />
Das Imputing funktioniert selbst bei 90%<br />
hochzurechnen<strong>de</strong>n SNP so gut, weil die<br />
drei Milliar<strong>de</strong>n genetischen Informationen,<br />
die je<strong>de</strong>s Tier zur Hälfte von Vater<br />
und Mutter bekommt, nicht wild gemixt,<br />
son<strong>de</strong>rn in größeren Teilabschnitten (Haplotypen)<br />
übertragen wer<strong>de</strong>n.<br />
ctga tgga gattac ccct aatga ctag gtagc ccct ctga<br />
tac ccct aatga ctag gtagc ccct ctga tgga gattac c<br />
ctag gtagc ccct ctga tgga gattac ccct aatga ctag g<br />
Der erste Schritt <strong>de</strong>s Imputing besteht<br />
also darin, die Teilabschnitte zu i<strong>de</strong>ntifizieren,<br />
die immer gemeinsam übertragen<br />
wer<strong>de</strong>n. Die verfügbaren Volltypisierungen<br />
in <strong>de</strong>r Population dienen dafür als<br />
eine Art Lernstichprobe. Die SNPs <strong>de</strong>r<br />
LD-Chips sind so ausgewählt, dass diese<br />
I<strong>de</strong>ntifizierung bestmöglich erfolgen kann,<br />
d.h., sie sind möglichst gleichmäßig über<br />
die Abschnitte verteilt.<br />
Anschließend wird in einem zweiten<br />
Schritt anhand <strong>de</strong>r typisierten 6.000 SNP<br />
festgestellt, aus welchen dieser gemeinsam<br />
übertragenen Teilstücke sich das<br />
Genom <strong>de</strong>s LD-typisierten Tieres zusammensetzt.<br />
Die innerhalb <strong>de</strong>r Teilstücke<br />
fehlen<strong>de</strong>n SNPs sollten dann i<strong>de</strong>ntisch<br />
mit <strong>de</strong>nen <strong>de</strong>r gleichen Teilabschnitte aus<br />
<strong>de</strong>r Lernstichprobe <strong>de</strong>r Volltypisierungen<br />
sein und wer<strong>de</strong>n entsprechend eingesetzt.<br />
Das Auffüllen <strong>de</strong>r Teilabschnitte lässt sich<br />
mit einem Puzzle vergleichen. Auch hier<br />
kann man oft anhand von nur wenigen,<br />
aber repräsentativ ausgewählten Teilchen<br />
das gesamte Bild z.B. eines Ferraris, erahnen<br />
und so <strong>de</strong>n fehlen<strong>de</strong>n Rest ergänzen.<br />
Das funktioniert aber nur, wenn man<br />
weiß, wie die aktuellen Ferrari-Mo<strong>de</strong>lle,<br />
bzw. die gemeinsam übertragenen Teilabschnitte,<br />
gera<strong>de</strong> aussehen. Deshalb wird<br />
die Lernstichprobe auch laufend durch<br />
die neuen, volltypisierten Besamungsbullen<br />
aktualisiert.<br />
tga tgga gattac ccct aatga ctag gtagc ccct ctga tg<br />
•<br />
Neuer EG-10K-LD-Chip macht<br />
genomische Untersuchung <strong>de</strong>utlich preisgünstiger<br />
c ccct aatga ctag gtagc ccct ctga tgga gattac ccct<br />
g gtagc ccct ctga tgga gattac ccct aatga ctag gta<br />
a tgga gattac ccct aatga ctag gtagc ccct ctga tgga<br />
Jetzt können Sie Ihre Nachzucht genomisch testen lassen. Mit <strong>de</strong>r Einführung <strong>de</strong>s neuen LD-Chip<br />
eröffnet sich eine neue günstige Möglichkeit <strong>de</strong>r Selektion weiblicher Tiere in Ihrer Her<strong>de</strong>.<br />
ccct aatga ctag gtagc ccct ctga tgga gattac ccct aa<br />
→<br />
→<br />
→<br />
→<br />
→<br />
→<br />
Sicheres Erkennen <strong>de</strong>r Stärken und Schwächen Ihrer Tiere<br />
Frühes Selektieren <strong>de</strong>r Tiere für die eigene Bestandsergänzung o<strong>de</strong>r <strong>de</strong>n Verkauf<br />
Gezieltere Anpaarung möglich<br />
Optimiertes Selektieren <strong>de</strong>r Tiere, die für <strong>de</strong>n Embryo-Transfer in Frage kommen<br />
steigen<strong>de</strong> Vermarktungschancen für Ihre männlichen und weiblichen Tiere<br />
Möglichkeit auf lukrative Anpaarungsverträge<br />
Das Gesamtpacket „Genotypisierung und genomische Zuchtwertschätzung“ mit <strong>de</strong>m LD-Chip kostet 75€.<br />
Bei weiteren Fragen Fragen können Sie sich gerne an Ihr CONVIS Milchrin<strong>de</strong>rteam wen<strong>de</strong>n!<br />
gtagc ccct gtagc ccct ctga tgga gattac ccct aatga<br />
ga gattac ccct aatga ctag gtagc ccct c<br />
Milchrin<strong>de</strong>r<br />
[passion for breeding]<br />
<strong>de</strong> <strong>lëtzebuerger</strong> ziichter 3|<strong>2013</strong>