Euroimmun Medizinische Labordiagnostika AG - Universität zu Lübeck
Euroimmun Medizinische Labordiagnostika AG - Universität zu Lübeck
Euroimmun Medizinische Labordiagnostika AG - Universität zu Lübeck
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
3. Ergebnisse 51<br />
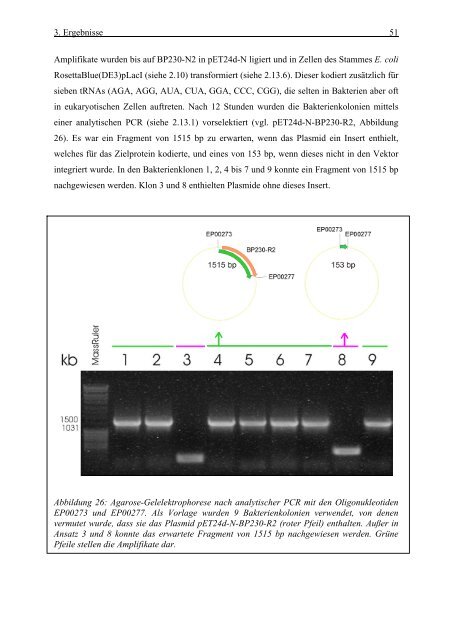
Amplifikate wurden bis auf BP230-N2 in pET24d-N ligiert und in Zellen des Stammes E. coli<br />
RosettaBlue(DE3)pLacI (siehe 2.10) transformiert (siehe 2.13.6). Dieser kodiert <strong>zu</strong>sätzlich für<br />
sieben tRNAs (<strong>AG</strong>A, <strong>AG</strong>G, AUA, CUA, GGA, CCC, CGG), die selten in Bakterien aber oft<br />
in eukaryotischen Zellen auftreten. Nach 12 Stunden wurden die Bakterienkolonien mittels<br />
einer analytischen PCR (siehe 2.13.1) vorselektiert (vgl. pET24d-N-BP230-R2, Abbildung<br />
26). Es war ein Fragment von 1515 bp <strong>zu</strong> erwarten, wenn das Plasmid ein Insert enthielt,<br />
welches für das Zielprotein kodierte, und eines von 153 bp, wenn dieses nicht in den Vektor<br />
integriert wurde. In den Bakterienklonen 1, 2, 4 bis 7 und 9 konnte ein Fragment von 1515 bp<br />
nachgewiesen werden. Klon 3 und 8 enthielten Plasmide ohne dieses Insert.<br />
Abbildung 26: Agarose-Gelelektrophorese nach analytischer PCR mit den Oligonukleotiden<br />
EP00273 und EP00277. Als Vorlage wurden 9 Bakterienkolonien verwendet, von denen<br />
vermutet wurde, dass sie das Plasmid pET24d-N-BP230-R2 (roter Pfeil) enthalten. Außer in<br />
Ansatz 3 und 8 konnte das erwartete Fragment von 1515 bp nachgewiesen werden. Grüne<br />
Pfeile stellen die Amplifikate dar.